试剂
reagent
产品简介:
简介:CUT&Tag是针对ChIP-Seq的局限性而创建的研究基因组中组蛋白修饰以及染色质结合蛋白和DNA相互作用的高效新技术。尽管与ChIP-Seq原理相似,CUT&Tag可以在新鲜细胞原生的状态下,利用Tn5转座酶复合体转座的特性在获取片段化DNA的同时完成了末端添加建库测序所需的扩增引物及条形码序列。它具备了操作简单快速 (1-2天可完成建库),无需交联固定和超声片段化,起始细胞数少(通常至少降低十倍以上),测序量和成本大大减少(通常降低五到十倍以上),信噪比好,重复性高等优势。
目前,该技术正在取代ChIP-Seq并越来越广泛地应用到基因组学和生物医学研究的各个方面。
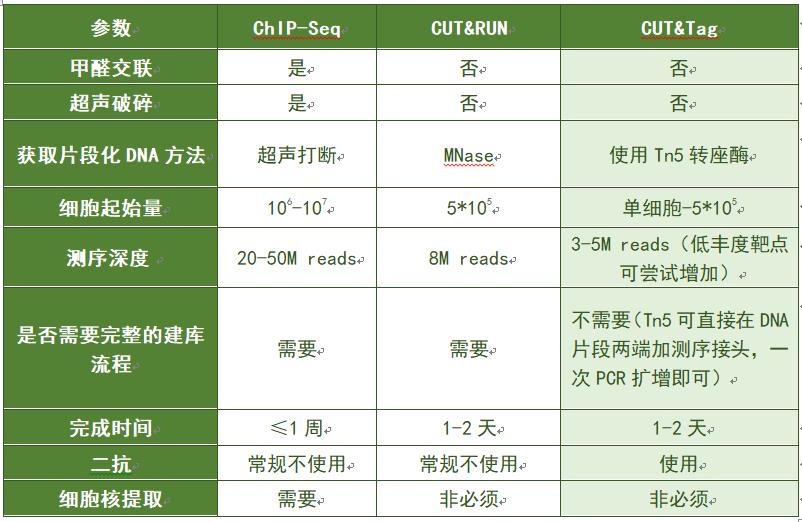
对比:相较于传统的ChIP-Seq方法,CUT&Tag无需甲醛交联和超声破碎,测序深度也大大减少,需要的细胞起始量更少(单细胞-5*1055),时间更短,质量更优的特点。因此,随着越来越多文献的发表,具有更优的性价比的CUT&Tag正在逐步替代ChIP-Seq。
应用方向:
CUT&Tag技术目前已经广泛应用于组蛋白修饰/染色质结合蛋白分析、开放染色质分析、单细胞组学、DNA甲基化分析、空间生物学和染色质构象捕获 (3C)等多种研究方向,并且适用于植物和动物的多种模式物种,在生物学中具有极其重要的研究意义!
实验方案设计:
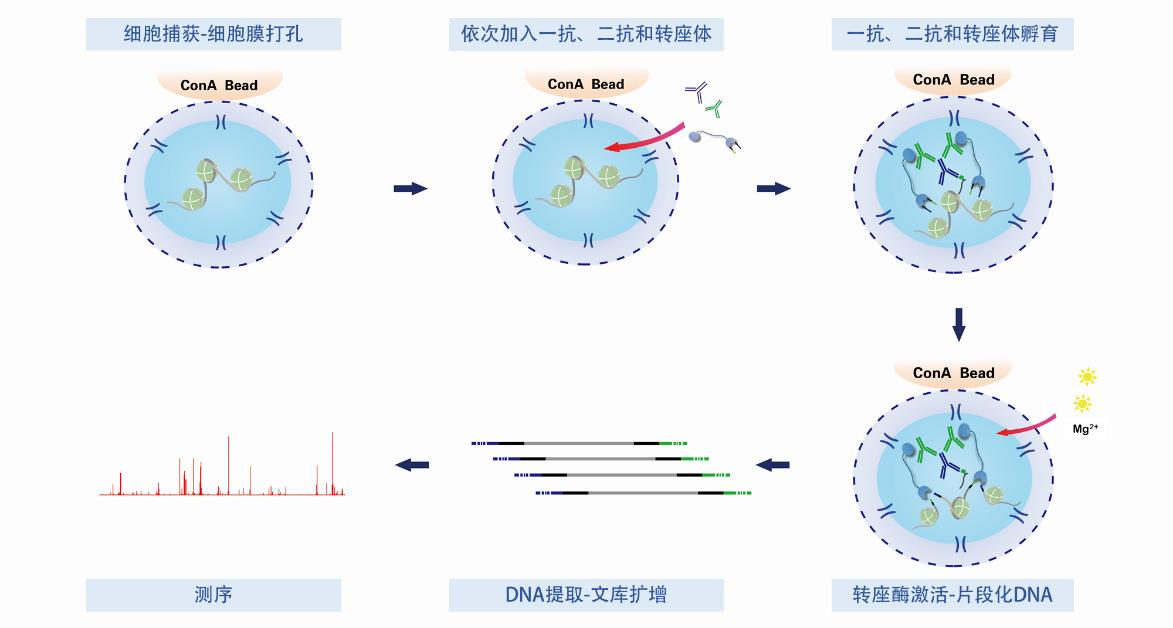
首先ConAbeads(ConcanavalinA-coated magnetic beads)与细胞膜或核膜相结合,随后通过穿孔剂对细胞进行穿孔处理,改变其通透性,依次加入和靶蛋白相对应的一抗、二抗和pA-Tn5Transposase进行孵育,再通过Mg2+激活Tn5酶,Tn5酶在激活的状态下会定向切割靶蛋白对应的DNA片段,同时在切割的DNA两端加上文库扩增的序列接头,以便于直接对回收的DNA片段进行PCR建库。随后文库质量检测合格后,便可直接上机测序。
实验流程简介:
* ConAbeads(ConcanavalinA-coated magnetic beads)与细胞膜或核膜相结合
* 一抗结合
* 二抗结合
* pA-Tn5Transposase结合
* Mg2+激活转座酶转座,从而实现DNA片段化
* 提取片段化的DNA
* PCR扩增建库和产物纯化
* 评估测量文库质量
实验流程图:
产品亮点总结:
细胞起始量较低(单细胞-5*105),从百万级降低到单细胞水平
结果重复性较高,且信噪比较高
把繁琐的ChIP流程缩短至1-2天的时间操作流程简单,无需甲醛交联、细胞破碎和超声片段化DNA
简化了文库构建流程,在转座子上添加了‘接头’,只需要简单的PCR即可获得高质量NGS文库
与ChIP相比,测序深度减少10倍左右
CUT&Tag与CUT&RUN 、 ChIP-Seq比对
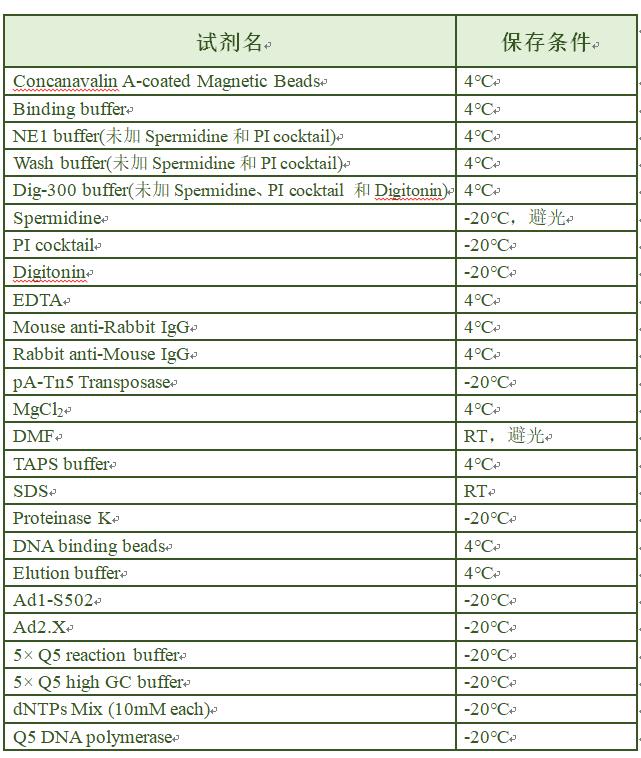
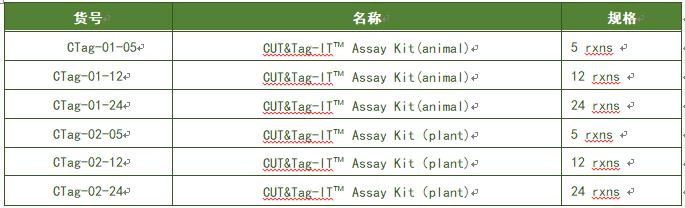
CUT&Tag-IT™ Assay Kit组成成分
本试剂盒各组成成分如下表所示,分为动物来源和植物来源两种试剂盒,请按照提示温度进行保存,有效期均为6个月。4℃储存的成分需要冰块运输,-20℃储存的成分需要干冰运输(尽量避免反复冻融)。
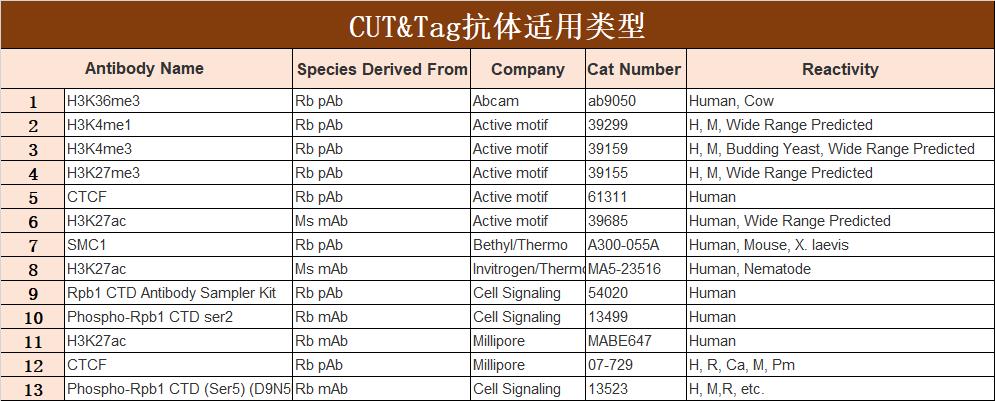
CUT&Tag验证过的适配抗体
目前,经过实验验证和参考发表的文献,整理出适配的靶点信息如下列表,如有任何疑问请咨询我们的技术人员。
CUT&Tag-IT™ Assay Kit 实验数据
1.H3K4me1抗体的CUT&Tag数据显示,与IgG对照组相比,H3K4me1 CUT&Tag检测到了H3K4me1的信号峰,且信噪比较高。
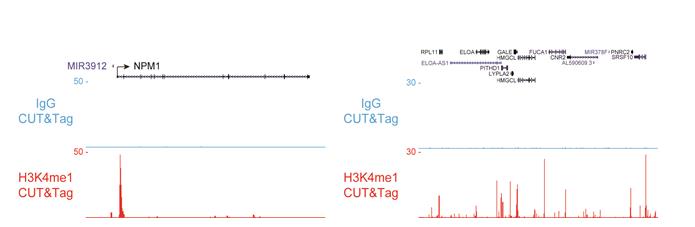
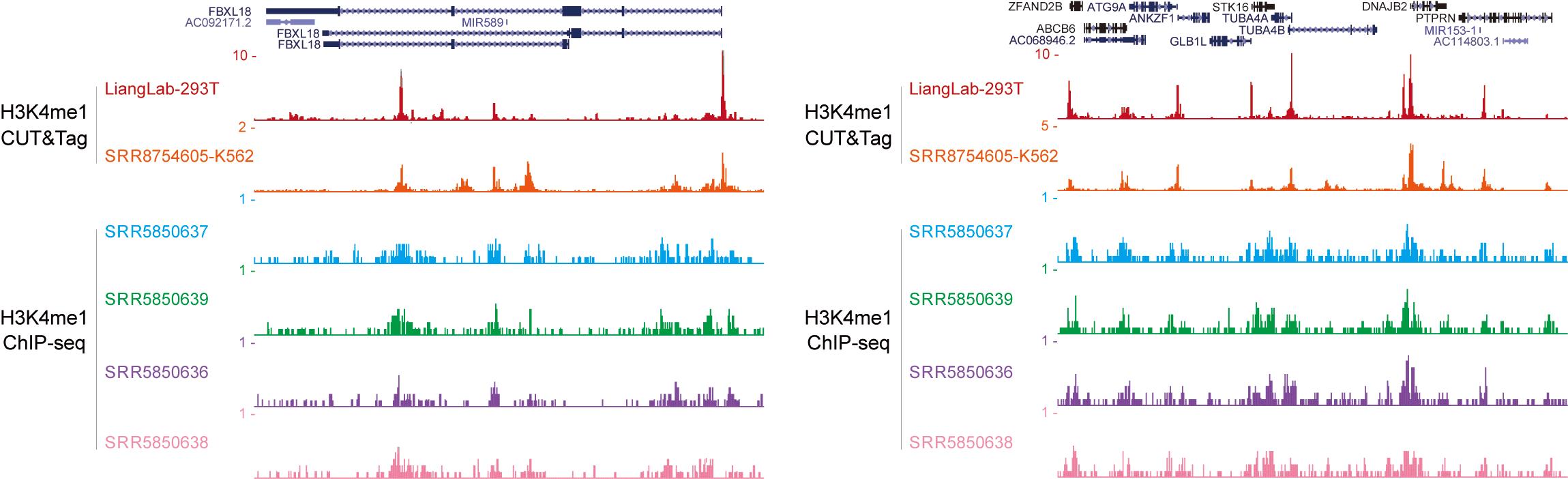
2.下图通过对比H3K4me1抗体的CUT&Tag数据与ChIP-seq数据,发现H3K4me1 CUT&Tag检测到H3K4me1的信号峰,并且信噪比较高。因此证明了CUT&Tag数据信噪比更高。
CUT&Tag-IT™ Assay Kit 问答部分
1.对于贴壁细胞样本,是否可以通过胰酶处理?
不可以,尽量使用器皿进行处理。胰酶可能对细胞膜通透性有影响。贴壁细胞刮下来,用非酶处理成单细胞。
2.操作说明书内的洋地黄皂苷用量是否适用于所有的细胞样本?
不一定,操作说明书内的用量是对于普通样本;对于一些不敏感的样本,需要先对样本进行洋地黄皂苷处理后,通过分析台盼蓝染色结果,来判断洋地黄皂苷用量。
3.样本最低使用量是多少?
目前实验数据使用量集中在105,更低的细胞量理论上也可使用。
4.是否可使用冷冻样本?
冷冻样本需要进行核提取,建议尽量使用新鲜样本。
5.对于植物细胞来说,CUT&Tag是否适用?
针对植物样本,我们设计了植物专用的操作说明书。
6.CUT&Tag和ChIP-Seq是否可以使用相同抗体?
不一定,ChIP-Seq目前已经验证过的抗体中,只有部分适合用于CUT&Tag,CUT&Tag已经验证过的抗体,请参照网页关于抗体选择部分的表格。
7.CUT&Tag和ChIP-Seq相比,优势是什么?
细胞起始量较低,从百万级降低到单细胞水平
结果重复性较高,且信噪比较高
把繁琐的ChIP流程缩短至1-2天的时间
操作流程简单,无需甲醛交联、细胞破碎和超声片段化DNA
无需文库构建流程
测序深度减少至少10-倍
8.阴性对照和阳性对照如何选择?
阴性对照我们推荐在实验过程中,去除一抗,只使用二抗(加一下二抗详细信息);阳性对照我们推荐H3K27me3(Rb pAb,Activemotif)。
9.如何评估最后构建的文库质量是否成功?
根据构建的文库片段长度判别,整体长度应该低于500bp。
10.CUT&Tag-IT™Assay Kit和ChIP-Seq数据分析方法是否一致?
一致
11.CUT&Tag构建的文库是否是双index ?
是的,测序时建议双向测序
CUT&Tag-IT™ Assay Kit商品
应用方向
1.组蛋白修饰/DNA 结合蛋白分析
使用 CUT&Tag 能够达到高效、低成本的组蛋白修饰分析,并且适用于不同类型的靶蛋白和转录因子。
Kaya-Okur H S , Janssens D H , Henikoff J G , et al. Efficient low-costchromatin profiling with CUT&Tag[J]. Nature Protocol, 2020.
2.单细胞组学
有关单细胞中基因表达和染色质可及性分析方法已经非常成熟,但是对于具有特定组蛋白修饰的染色质区域进行单细胞分析仍具有很大的挑战性。传统的ChIP-Seq需要百万数量级的细胞样本,而CUT&Tag 技术可用于单细胞测序平台,在肿瘤研究方向上应用较多,例如通过CUT&Tag对脑肿瘤患者治疗前后的 H3K27me3 进行分析,来确定肿瘤微环境中的细胞类型以及原始样本和治疗后 PcG 活性的异质性。
Wu S J , Furlan S N , Mihalas A B , et al.Single-cell CUT&Tag analysis of chromatin modifications in differentiationand tumor progression[J]. Nature Biotechnology.
3.DNA甲基化分析
目前使用有限的细胞样本来破译 DNA 甲基化、组蛋白修饰和DNA 序列之间的复杂关系仍然是一个挑战。但是通过CUT&Tag技术可以在单个实验中达到单碱基分辨率,同时分析DNA 的甲基化和组蛋白修饰的基因组定位。
Li R , Grimm S A , Wade P A . CUT&Tag-BS for simultaneous profiling of histonemodification and DNA methylation with high efficiency and low cost -ScienceDirect[J]. 2021.
4.空间生物学
空间组学成为生物学和生物医学研究的新前沿。 CUT&Tag可用于空间分辨的全基因组组蛋白修饰分析。例如小鼠胚胎中空间分辨的染色质状态揭示了与 ENCODE 参考一致的组织类型特异性表观遗传调控,并提供了组织尺度的空间信息。组织和细胞水平的空间分辨的染色质修饰分析将为正常生理和发病机制中的表观遗传调控、细胞功能研究提供新方法。
Deng Y, Bartosovic M, Kukanja P, Zhang D, LiuY, Su G, Enninful A, Bai Z, Castelo-Branco G, Fan R. Spatial-CUT&Tag:Spatially resolved chromatin modification profiling at the cellular level.Science. 2022 Feb 11;375(6581):681-686. doi: 10.1126/science.abg7216. Epub 2022Feb 10. PMID: 35143307.
5.染色质构象捕获 (3C)
基因组的三维结构对于研究基因表达调控机制至关重要,通过将CUT&Run技术与传统的染色质构象捕获技术(3C、Hi-C)相结合,可以进一步的识别和分析以蛋白为中心的染色质构象,用于长距离相互作用和染色质三维结构研究,在空间结构中的研究具有重大意义。
Shimbo T , Kawamura M , Wijaya E , et al.Cut-C: cleavage under tethered nuclease for conformational capture[J]. BMCGenomics, 2019, 20(1).


